摘要:血细胞智能检测与计数软件应用深度学习技术智能检测血细胞图像中红细胞、镰状细胞等不同形态细胞并可视化计数,以辅助医学细胞检测。本文详细介绍血细胞智能检测与计数软件,在介绍算法原理的同时,给出Python的实现代码以及PyQt的UI界面。在界面中可以选择各种图片、视频进行检测识别;可对图像中存在的多目标进行识别分类,检测速度快、识别精度高。博文提供了完整的Python代码和使用教程,适合新入门的朋友参考,完整代码资源文件请转至文末的下载链接。本博文目录如下:
前言
目标检测主要通过人工智能技术识别并定位图像中的物体,在实际生活中应用领域广泛:数字摄像机智能火灾监控、医学影像肿瘤检测、数码相机人脸自动定位等等多个领域。传统目标检测算法因无法利用图像的深层特征而易受到物体遮挡、光照变化等因素的干扰,导致漏检与误检。深度学习的出现可以很好地解决这一问题。深度学习算法可以从样本中学习,通过对特征的加工组合,进而提取图像的更深层特征。本文在深度学习理论的支持下研究目标检测在血液细胞中的应用。。
医疗图像的获取以及标注需要耗费巨大的人力成本,而训练数据不足会导致模型出现过拟合的现象,尤其是血液细胞中各类细胞的识别。并且血液细胞图像本身存在着对比度低和各类细胞数量、外形差异较大的特点,这些都影响着血液细胞检测的精度。本文在对深度学习目标检测算法研究的基础上,对血液细胞中的各类细胞识别,并使用算法得到较高的血液细胞的检测精度。
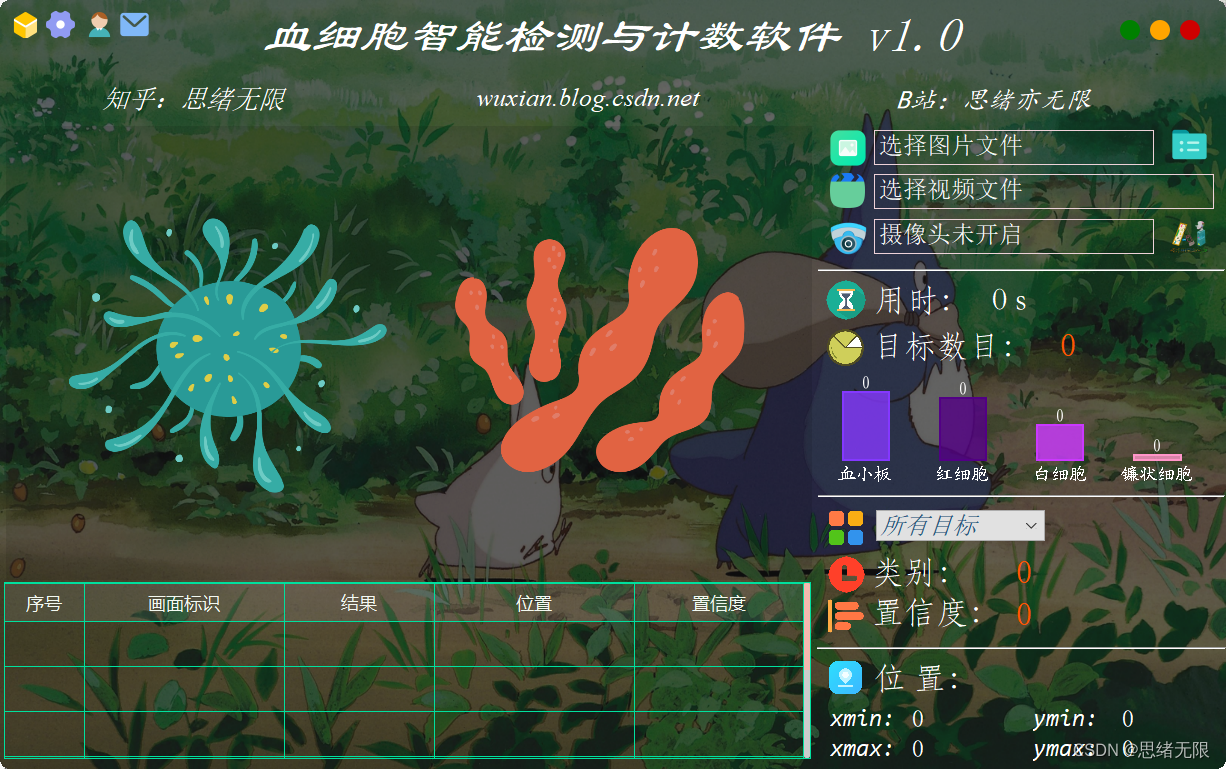
本系统采用登录注册进行用户管理,对于图片、视频和摄像头捕获的实时画面,可检测医疗图像,系统支持结果记录、展示和保存,每次检测的结果记录在表格中。对此这里给出博主设计的界面,同款的简约风,功能也可以满足图片、视频和摄像头的识别检测,希望大家可以喜欢,初始界面如下图:
检测类别时的界面截图(点击图片可放大)如下图,可识别画面中存在的多个类别,也可开启摄像头或视频检测:
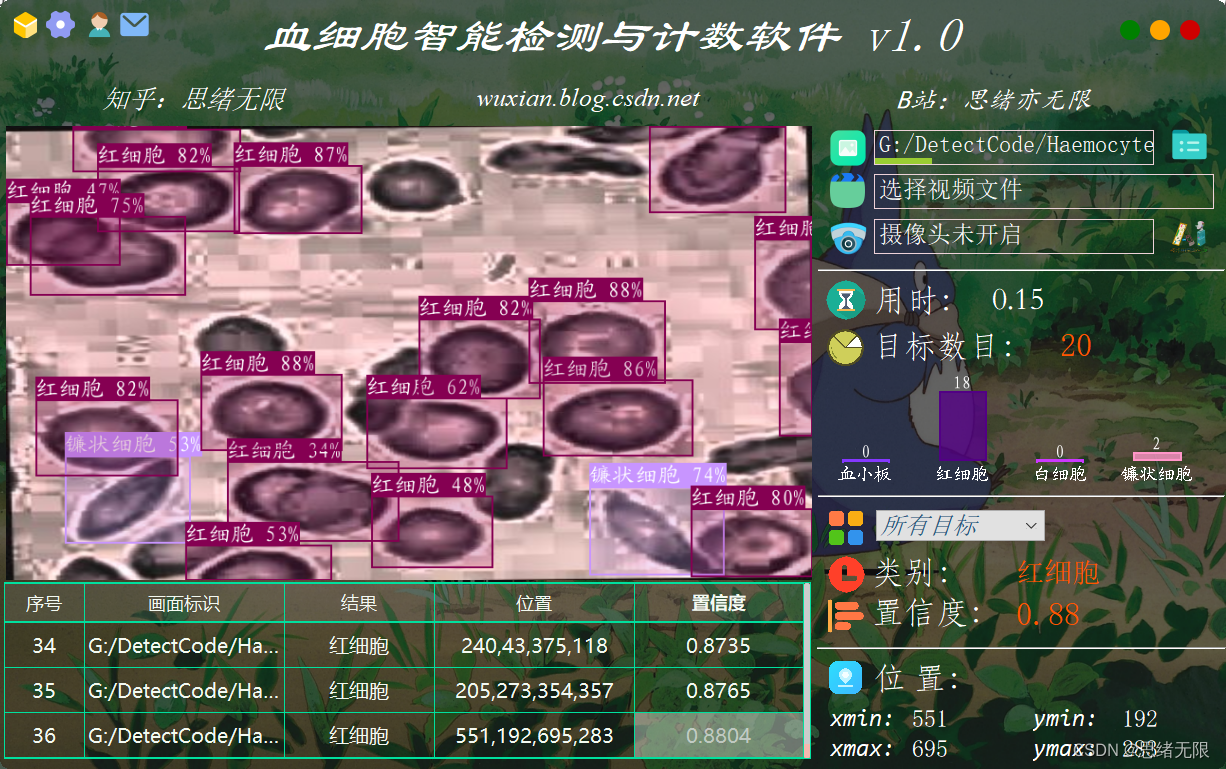
详细的功能演示效果参见博主的B站视频或下一节的动图演示,觉得不错的朋友敬请点赞、关注加收藏!系统UI界面的设计工作量较大,界面美化更需仔细雕琢,大家有任何建议或意见和可在下方评论交流。
1. 效果演示
软件好不好用,颜值很重要,首先我们还是通过动图看一下识别的效果,系统主要实现的功能是对图片、视频和摄像头画面中的血细胞进行识别,识别的结果可视化显示在界面和图像中,另外提供多个目标的显示选择功能,演示效果如下。
(一)系统介绍
血细胞智能检测与计数软件主要用于显微镜下的血细胞检测与计数,基于深度学习技术识别图像中常见的4种血细胞,包括血小板、红细胞、白细胞、镰状细胞等,输出细胞的标记框坐标和类别,以辅助自动化细胞统计和医学研究;软件提供登录注册功能,可进行用户管理;软件能够有效识别电子显微镜采集的细胞图片、视频等文件形式,检测各种细胞形态,并记录识别结果在界面表格中方便查看;可开启摄像头实时监测和统计当前视野范围各种类型细胞数目,支持结果记录、展示和保存。
(二)技术特点
(1)YoloV5目标检测算法检测血细胞,模型支持更换;
(2)摄像头实时检测血细胞,展示、记录和保存结果;
(3)检测图片、视频等图像中的血细胞个体;
(4)支持用户登录、注册,检测结果可视化功能;
(三)用户注册登录界面
这里设计了一个登录界面,可以注册账号和密码,然后进行登录。界面还是参考了当前流行的UI设计,左侧是一个动图,右侧输入账号、密码、验证码等等。

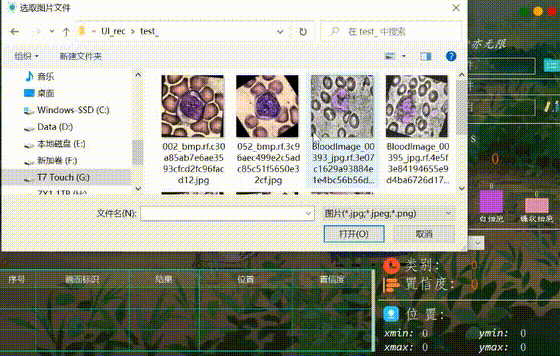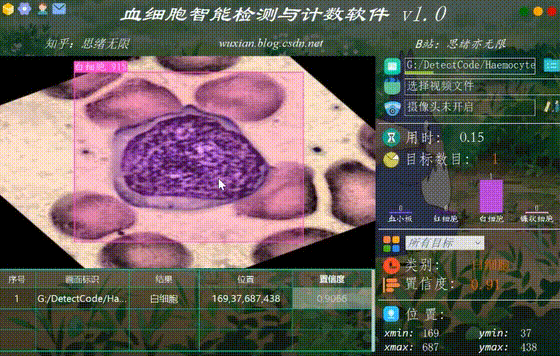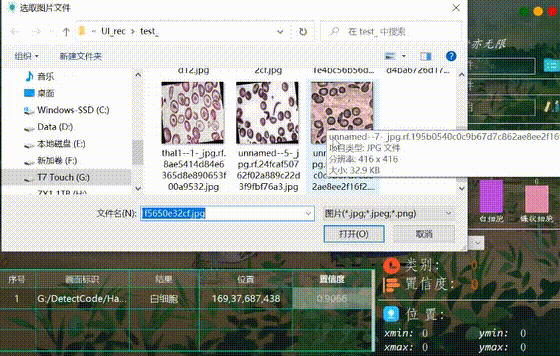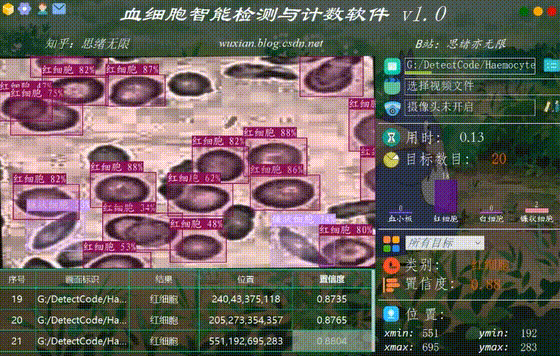
(四)选择图片识别
系统允许选择图片文件进行识别,点击图片选择按钮图标选择图片后,显示所有识别的结果,可通过下拉选框查看单个结果,以便具体判断某一特定目标。本功能的界面展示如下图所示:
(五)视频识别效果展示
很多时候我们需要识别一段视频中的血细胞,这里设计了视频选择功能。点击视频按钮可选择待检测的视频,系统会自动解析视频逐帧识别多个血细胞,并将血细胞的分类和计数结果记录在右下角表格中,效果如下图所示:
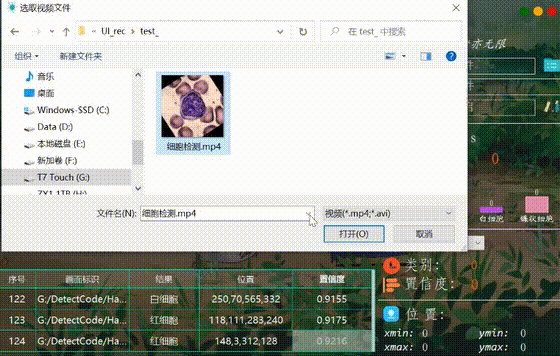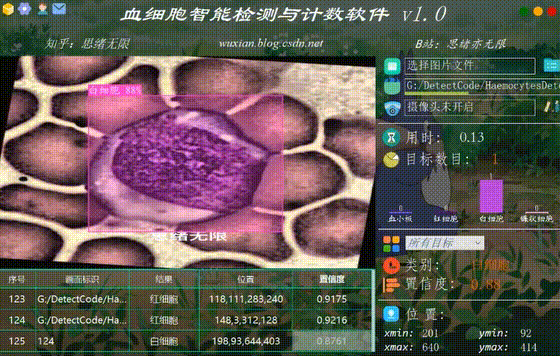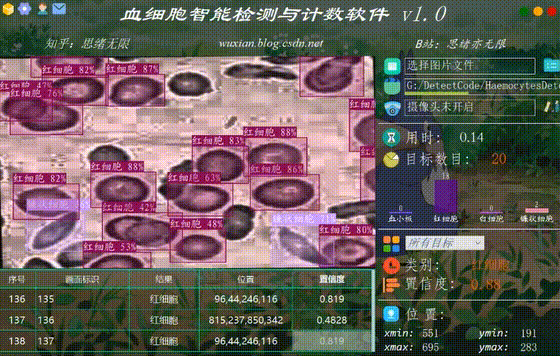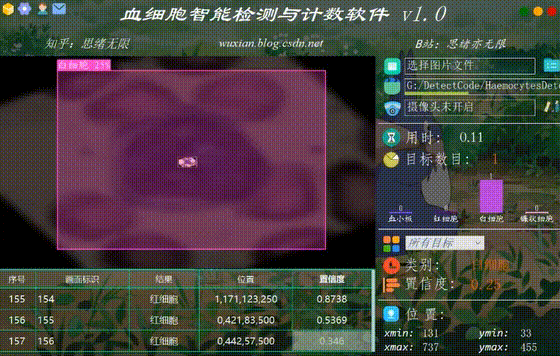
(六)摄像头检测效果展示
在真实场景中,我们往往利用摄像头获取实时画面,同时需要对血细胞进行识别,因此本文考虑到此项功能。如下图所示,点击摄像头按钮后系统进入准备状态,系统显示实时画面并开始检测画面中的血细胞,识别结果展示如下图:

2. 血细胞数据集和模型训练
(一)数据集制作
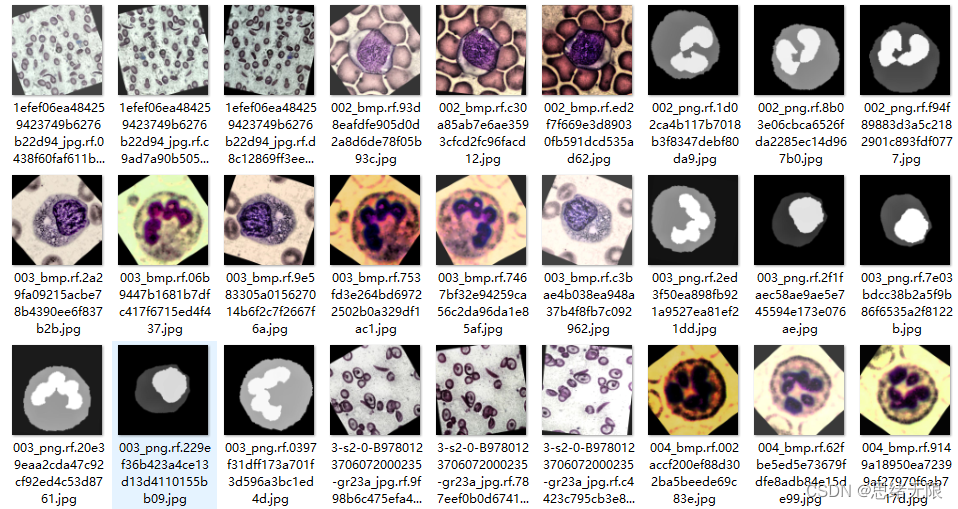
本文实验的血细胞数据集包含训练集2853张图片,验证集219张图片,测试集81张图片,共计3153张图片,选取部分数据部分样本数据集如图所示。
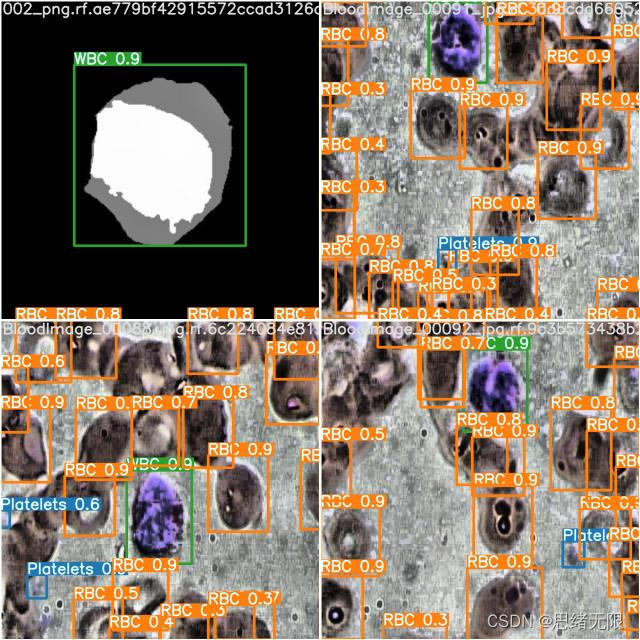
每张图像均提供了图像类标记信息,图像中细胞的bounding box,目标的属性信息,数据集并解压后得到如下的图片
船舰数据集的类别信息如下,包含血小板、红细胞、白细胞、镰状细胞等类别
Chinese_name = {'Platelets': "血小板", 'RBC': "红细胞", 'WBC': "白细胞", 'sickle cell': "镰状细胞"}- 1
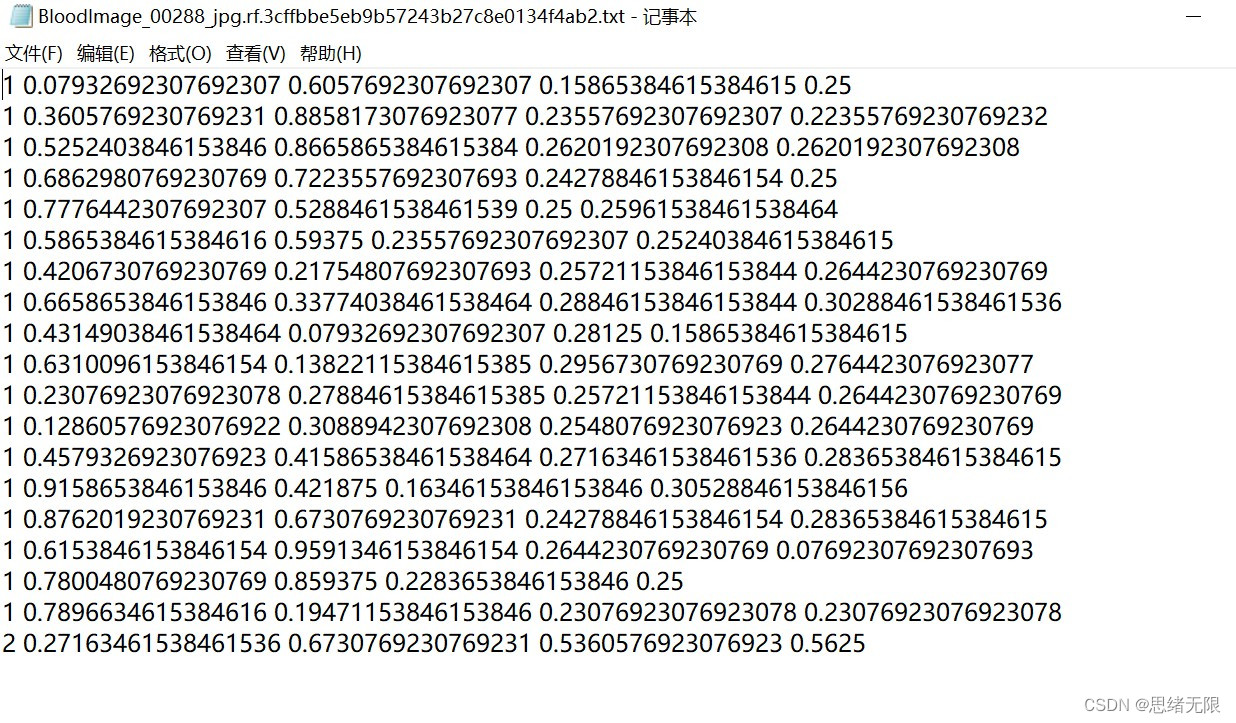
原数据格式是xml文件对目标细胞注释,现在需要将这种注释转换为yolov5所需的格式。即每个图像对应一个txt文件,文件中存储该图像中全部细胞的类别和坐标,一行存储一个细胞的信息,如下图
在本项目的同级目录下创建数据集存储路径,路径内创建训练集,测试集路径和配置文件dataset.yaml
images内存储图像数据,labels存储标注数据,文件名称对应相同,设置数据集配置文件如下
train: ./Haemocytes/images/train
val: ./Haemocytes/images/valid
test: ./Haemocytes/images/test
nc: 4
names: ['Platelets', 'RBC', 'WBC', 'sickle cell'] (1)train 指定训练集图像路径
(2)val 指定验证集图像路径
(3)nc 指定目标类别数量 这里为血小板,红细胞,白细胞共3种
(4)目标对应类别名称
接下来需要配置项目路径内的train.py的相关参数,指定预训练的模型权重和模型结构文件和数据集配置文件,训练的轮数,batch_size的大小和输入图像的分辨率。
parser = argparse.ArgumentParser()
parser.add_argument('--weights', type=str, default='yolov5s.pt', help='initial weights path')
parser.add_argument('--cfg', type=str, default='models/yolov5s.yaml', help='model.yaml path')
parser.add_argument('--data', type=str, default='dataset.yaml', help='data.yaml path')
parser.add_argument('--hyp', type=str, default='data/hyp.scratch.yaml', help='hyperparameters path')
parser.add_argument('--epochs', type=int, default=300)
parser.add_argument('--batch-size', type=int, default=1, help='total batch size for all GPUs')
parser.add_argument('--img-size', nargs='+', type=int, default=[640, 640], help='[train, test] image sizes') 在我们的训练过程中,mAP50作为一种常用的目标检测评估指标很快达到了较高水平,而mAP50:95也在训练的过程中不断提升,说明我们模型从训练-验证的角度表现良好。读入一个测试文件夹进行预测,通过训练得到的选取验证集上效果最好的权重best.pt进行实验,得到PR曲线如下图所示。
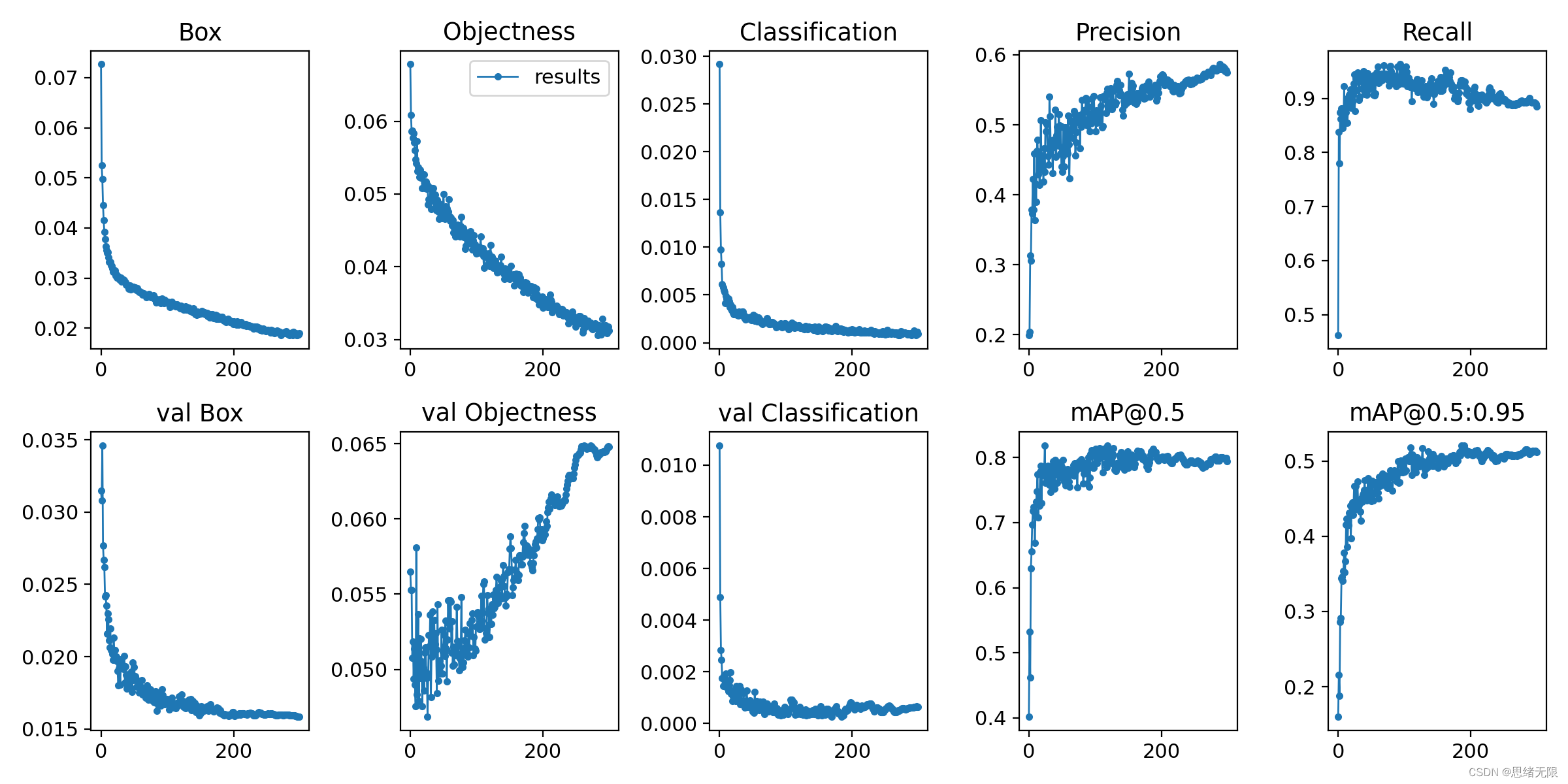
在深度学习中,我们通常通过损失函数下降的曲线来观察模型训练的情况。而YOLOv5训练时主要包含三个方面的损失:矩形框损失(box_loss)、置信度损失(obj_loss)和分类损失(cls_loss),在训练结束后,我们也可以在logs目录下找到生成对若干训练过程统计图。下图为博主训练血细胞识别的模型训练曲线图。
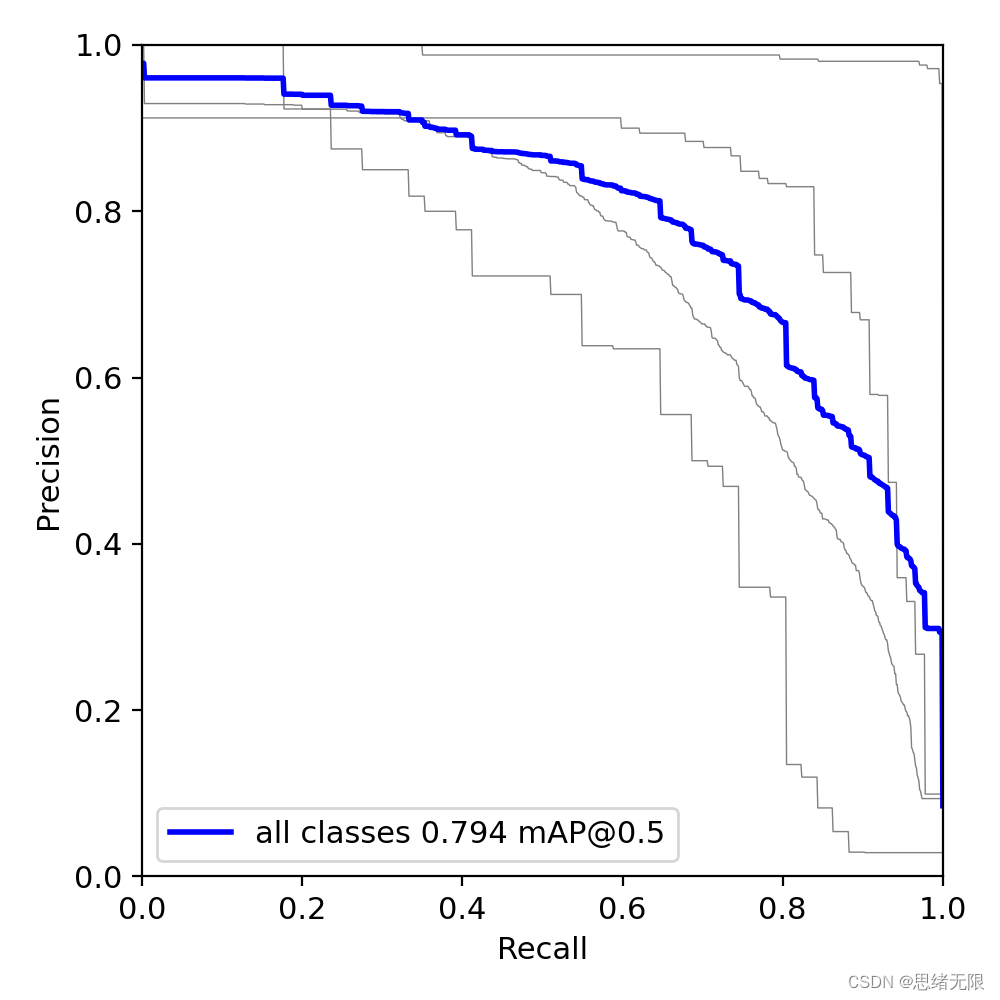
以PR-curve为例,可以看到我们的模型在验证集上的均值平均准确率为0.794。
3. 血细胞检测识别
运行testVideo得到预测结果,我们便可以将帧图像中的血细胞框出,然后在图片上用opencv绘图操作,输出血细胞的类别及血细胞的预测分数。以下是读取血细胞视频并进行检测的脚本,首先将图片数据进行预处理后送predict进行检测,然后计算标记框的位置并在图中标注出来。
while True:
# 从视频文件中逐帧读取画面
(grabbed, image) = vs.read()
# 若grabbed为空,表示视频到达最后一帧,退出
if not grabbed:
print("[INFO] 运行结束...")
output_video.release()
vs.release()
exit()
# 获取画面长宽
if W is None or H is None:
(H, W) = image.shape[:2]
image = cv2.resize(image, (850, 500))
img0 = image.copy()
img = letterbox(img0, new_shape=imgsz)[0]
img = np.stack(img, 0)
img = img[:, :, ::-1].transpose(2, 0, 1) # BGR to RGB, to 3x416x416
img = np.ascontiguousarray(img)
pred, useTime = predict(img)
det = pred[0]
p, s, im0 = None, '', img0
if det is not None and len(det): # 如果有检测信息则进入
det[:, :4] = scale_coords(img.shape[1:], det[:, :4], im0.shape).round() # 把图像缩放至im0的尺寸
number_i = 0 # 类别预编号
detInfo = []
for *xyxy, conf, cls in reversed(det): # 遍历检测信息
c1, c2 = (int(xyxy[0]), int(xyxy[1])), (int(xyxy[2]), int(xyxy[3]))
# 将检测信息添加到字典中
detInfo.append([names[int(cls)], [c1[0], c1[1], c2[0], c2[1]], '%.2f' % conf])
number_i += 1 # 编号数+1
label = '%s %.2f' % (names[int(cls)], conf)
# 画出检测到的目标物
plot_one_box(image, xyxy, label=label, color=colors[int(cls)])
# 实时显示检测画面
cv2.imshow('Stream', image)
image = cv2.resize(image, (vw, vh))
output_video.write(image) # 保存标记后的视频
if cv2.waitKey(1) & 0xFF == ord('q'):
break
# print("FPS:{}".format(int(0.6/(end-start))))
frameIndex += 1执行得到的结果如下图所示,图中血细胞的种类和置信度值都标注出来了,预测速度较快。基于此模型我们可以将其设计成一个带有界面的系统,在界面上选择图片、视频或摄像头然后调用模型进行检测。

博主对整个系统进行了详细测试,最终开发出一版流畅得到清新界面,就是博文演示部分的展示,完整的UI界面、测试图片视频、代码文件,以及Python离线依赖包(方便安装运行,也可自行配置环境),均已打包上传,感兴趣的朋友可以通过下载链接获取。
下载链接
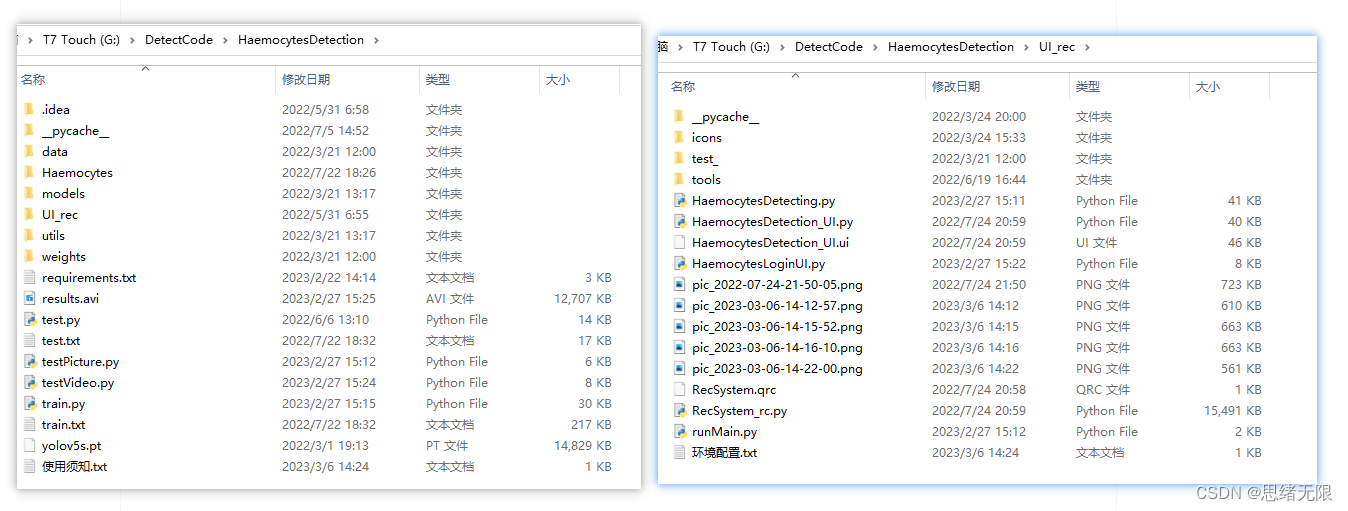
若您想获得博文中涉及的实现完整全部程序文件(包括测试图片、视频,py, UI文件等,如下图),这里已打包上传至博主的面包多平台,见可参考博客与视频,已将所有涉及的文件同时打包到里面,点击即可运行,完整文件截图如下:
在文件夹下的资源显示如下,下面的链接中也给出了Python的离线依赖包,读者可在正确安装Anaconda和Pycharm软件后,复制离线依赖包至项目目录下进行安装,离线依赖的使用详细演示也可见本人B站视频:win11从头安装软件和配置环境运行深度学习项目、Win10中使用pycharm和anaconda进行python环境配置教程。
注意:该代码采用Pycharm+Python3.8开发,经过测试能成功运行,运行界面的主程序为runMain.py和LoginUI.py,测试图片脚本可运行testPicture.py,测试视频脚本可运行testVideo.py。为确保程序顺利运行,请按照requirements.txt配置Python依赖包的版本。Python版本:3.8,请勿使用其他版本,详见requirements.txt文件;
完整资源中包含数据集及训练代码,环境配置与界面中文字、图片、logo等的修改方法请见视频,项目完整文件下载请见参考博客文章里面,或参考视频的简介处给出:➷➷➷
参考博客文章:https://zhuanlan.zhihu.com/p/615303348
参考视频演示:https://www.bilibili.com/video/BV1p84y1A7Yj/
离线依赖库下载链接:https://pan.baidu.com/s/1hW9z9ofV1FRSezTSj59JSg?pwd=oy4n (提取码:oy4n )
界面中文字、图标和背景图修改方法:
在Qt Designer中可以彻底修改界面的各个控件及设置,然后将ui文件转换为py文件即可调用和显示界面。如果只需要修改界面中的文字、图标和背景图的,可以直接在ConfigUI.config文件中修改,步骤如下:
(1)打开UI_rec/tools/ConfigUI.config文件,若乱码请选择GBK编码打开。
(2)如需修改界面文字,只要选中要改的字符替换成自己的就好。
(3)如需修改背景、图标等,只需修改图片的路径。例如,原文件中的背景图设置如下:
mainWindow = :/images/icons/back-image.png可修改为自己的名为background2.png图片(位置在UI_rec/icons/文件夹中),可将该项设置如下即可修改背景图:
mainWindow = ./icons/background2.png